|
|
|
研究は主として、バイオ系、IT系の2つで実施した。以下に主な成果を示す。
・ 研究概要:バイオ系
- 進化的計算手法を用いた遺伝子ネットワーク推定システムの設計・開発
遺伝子間の相互作用(遺伝子ネットワーク)の推定は、DNA マイクロアレイなどから得られた発
現量のタイムコースデータから、これを再現する数理モデルを推定することで行った。これまで
に、数理モデルにべき乗則に基づくS-system 表記の連立微分方程式、数理モデルを最適化する
数値最適化手法に遺伝的アルゴリズムを用いた手法が研究・開発されている。しかし、S-system
表記の微分方程式は、ある物質の生成・分解のプロセスをそれぞれ一つの項で表現する近似式で
あるため、ネットワーク構造の概略は得られるものの、詳細な構造は分からないという問題が生
じる。そこで、より詳細なネットワーク構造の表現が可能な数理モデルとして、質量作用則に基
づいた連立微分方程式表記を採用し、微分方程式の構造自体を最適化するための最適化手法とし
て遺伝的プログラミングを用いた手法を提案し、与えたデータ系列から相互作用を示す関数を自
動的に推定するシステムを設計・開発した。
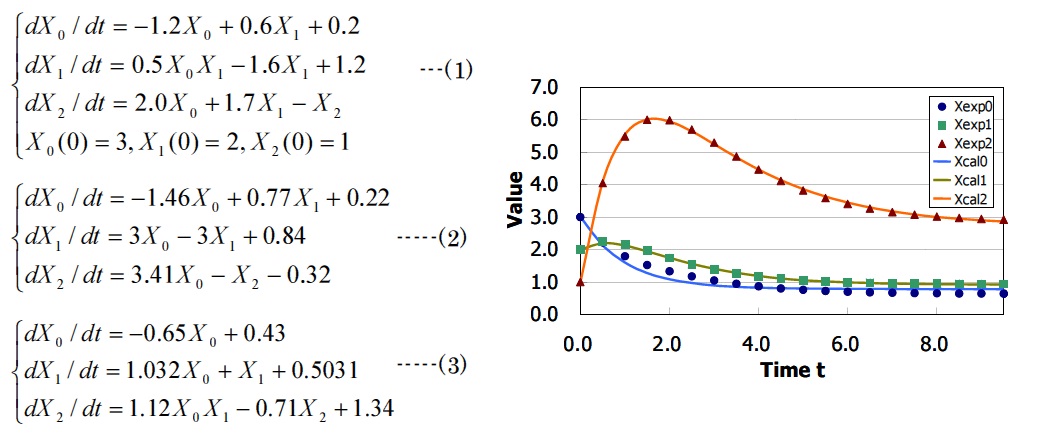
具体的には、システムの性能を評価するためのケーススタディとして式(1)を設定し、式(1)を
Runge-Kutta 法でサンプリングポイント0.5 (0≦t<10)で解いて得られた各変数20 点のデータを
データ系列として与え、元の式(式(1))が推定できるか調べた。その結果、50 試行中9 試行のみ最
適化が成功し、最良および最悪の結果で導出された方程式は式(2)および式(3)となり、成功率も
低く最良の場合でも元の式とはかなり異なる式しか得ることはできなかった。しかし、与えた
データ系列(Xexp)および得られたデータ系列(Xcal, 最悪の結果(式(3))をグラフで表すと下図の
ようになり、得られた式は元の式とは異なるがデータ系列は良く似ていることがわかった。こ
れは、与えたデータ系列が一つであったために拘束条件が非常に少なく、与えたデータ系列を
再現する方程式が多く存在し、一意に決定することができなかったことが要因と言える。
- マルチプルアラインメントアルゴリズムの開発および精度評価方法の改良
2006 年度は、2005 年度の時よりも大規模なベンチマークを行い、我々の方法がペアワイズ
アラインメント情報を用いた世界最高精度を誇る方法と遜色無い精度であることがわかった。
また、これまでに実装してきたプログラムを整備し、PRIME というプログラムとしてGPLで
公開した。PRIME はhttp://prime.cbrc.jp/で公開しているが、論文(Yamada,S. et al ., BMC
Bioinformatics 7:524)のSupplementary Data としても入手可能である。現在はPRIME を高
速化するためのヒューリスティックの導入と評価を行っている最中であるが、現時点では精度
の低下を1,2%に抑えながらも、約2 倍の高速化を実現した。
- タンパク質ドメインリンカー予測手法の高精度化
タンパク質の立体構造解析を精度よく行うためには、タンパク質を構成するドメインに相当
する領域を決定する必要がある。ドメイン領域が正確に予測できれば、各ドメインに分割して
実験することで、タンパク質の立体構造解析が容易になることが期待できる。
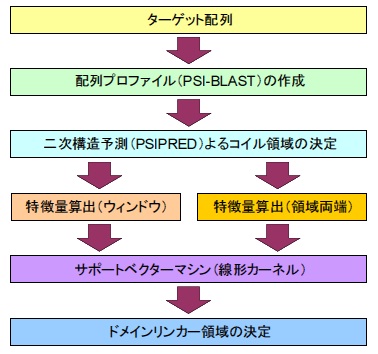
ドメイン領域は長くて多様性に富んでおり、直接予測
するのが困難であるため、本研究では、ドメイン間をつ
なぐループ領域であるドメインリンカー領域を予測す
る、というアプローチをとった。具体的には、配列プロ
ファイルを利用して位置依存性を考慮した予測手法を
提案した(右図)。
評価実験として、106 個のマルチドメインタンパク質
からなるデータセットを用いてジャックナイフテスト
を行った。その結果、従来手法に比べ、Sensitivity で約
5〜15%、Specificity で約40〜50%程度の精度が向上し
た。
・ 研究概要:IT系
- 商用サーチエンジンのランキング調査
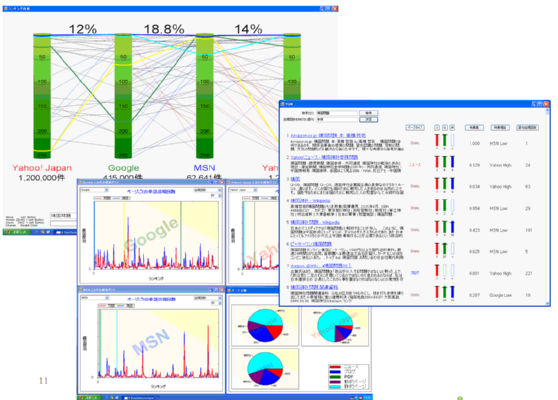
本研究所が得意とする解析技術を生かし、複数の商用サーチエンジンのランキングをユーザ自身が解析できる仕組みとして、下図に示すツールを作成すると共に、Google、Yahoo!JAPAN、MSNのランキングについて調査を行った。その結果、ランキングは毎日大きく変動すると共に、検索エンジンによって、強い分野、弱い分野があることが判明した。具体的にはニュース系はGoogleが強く、静的なWebページはYahoo!JAPANが得意とすることがわかった。
- 改変画像検知手法の提案
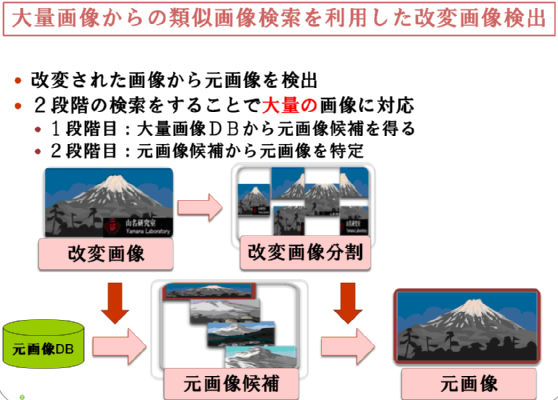
本研究所が得意とする解析技術を画像解析に生かすため、著作権違反となる画像検知を目的として、改変画像検知手法の提案を行った。主たるポイントは、下図に示す通り2段階検索を採用することで、50万枚レベルで数秒での検出を可能とした点である。(新聞報道有)
・ 講演会等
- 2006年11月7日 ボン大学との研究交流会 場所:早大理工55号館第1会議室
- 2006年6月20日 見学会 場所:産総研臨海副都心センター別館
・ 受託研究
- (株)富士通総研より「商用サーチエンジンのランキング調査」(経済産業省「次世代の知的情報アクセスに関する調査研究事業」における調査・研究の一環)
- (株)三菱総合研究所より「情報大航海プロジェクト共通技術及びFAST ESP に関する調査」(経済産業省「情報大航海18 年度先行調査・開発」の一環)
・その他の活動
- 連携大学院開始への貢献
2006 年3 月24 日にIT バイオ研究所メンバーが中心となり「早稲田大学理工学術院と独立行政
法人産業技術総合研究所との教育研究協力に関する協定書」を早稲田と産総研との間で締結。2006
年度より連携大学院方式に基づく、教育・研究を実施している。
連携大学院方式による講座
「バイオインフォマティクス特論」(2006年度〜(2010 年度も継続中))
担当者 (ITバイオ研究所 客員研究員のメンバー):
秋山泰・諏訪牧子・野口保・藤渕航・福田堅一郎・長野希美・関嶋政和・本野千恵・塚本弘毅・
向井有理
シラバス:
バイオインフォマティクス分野の重要なトピックスに関する講義を行う。
また実際の生物データに関する情報処理などを生命情報科学研究センター内で実習する。
成績評価は講義への出席状況および学生による研究成果報告により行なう。扱うトピックス例は
次のとおり。
- タンパク質構造変化のバイオインフォマティックス(野口)
- ゲノム解析(藤渕)
- 医学生物学知識の記号処理・自然言語処理(福田)
- 膜タンパク質のバイオインフォマティックス(諏訪. 向井)
- 分子動力学法による耐熱性タンパク質の解析(本野)
- 大規模生体分子シミュレーションとプリオンタンパク質の機能解析(関嶋)
- 酵素触媒機構の解析(長野)
- バイオインフォマティクスにおける大規模並列計算技術(秋山. 野口)
- 創薬のための大規模分子動力学および量子化学計算手法(塚本)
・論文等はこちら⇒ 2006年度論文.pdf
|


